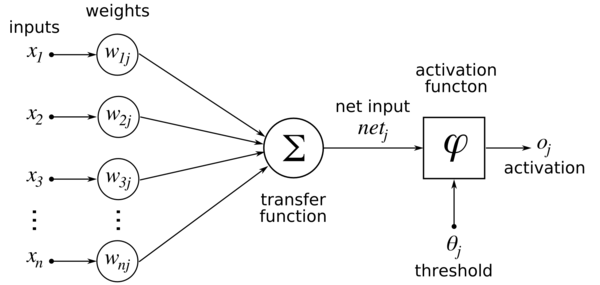
Een vorm van een gelaagd neuraal netwerk (perceptron) (afb: WikiMedia Commons)
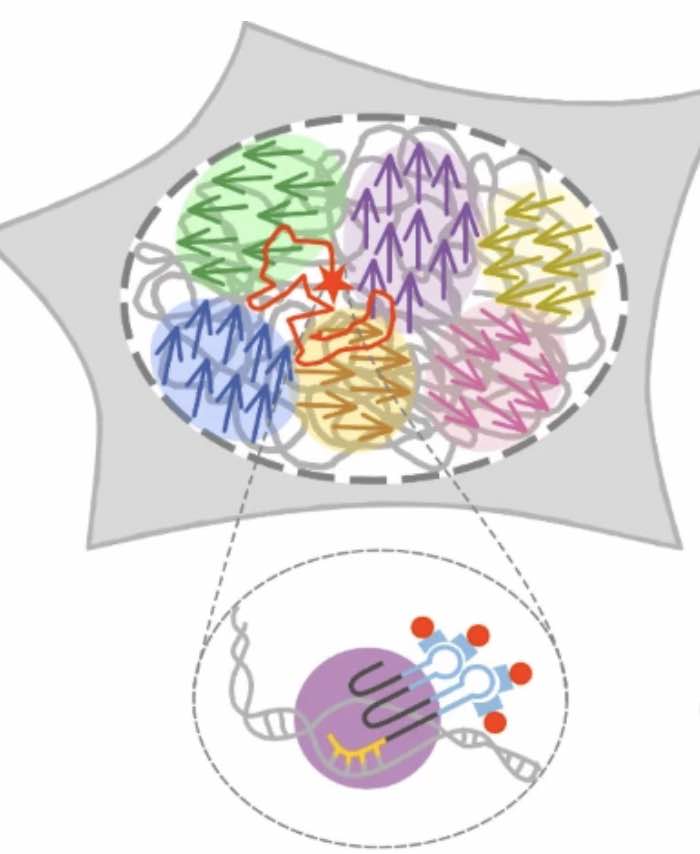
Onderzoekers in China en de VS hebben een neuraal netwerk van eiwitten in levende cellen ‘geconstrueerd’ dat verschillende signalen kan verwerken en op basis daarvan beslissingen kan nemen zoals ‘val dood’. Ze hebben dat percepteïne gedoopt, een knutselwoord bestaand uit delen van proteïne (eiwit) en perceptron (een gelaagd neuraal netwerk). Ik(=as) moet zeggen dat ik nog niet meteen goed weet wat ze daarmee willen doen, maar ik laat me graag verrassen… Lees verder