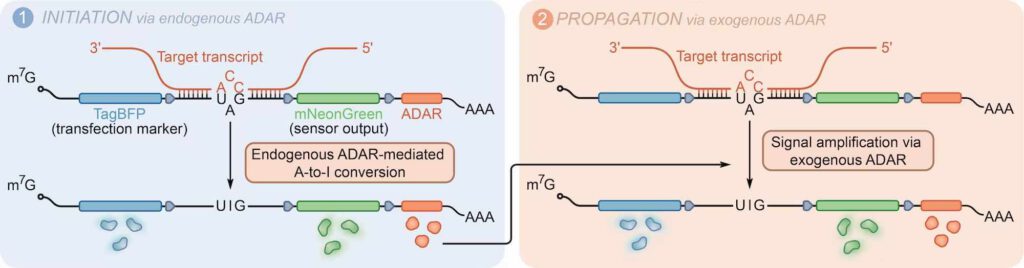
Het proces in beeld. Als A in I wordt omgezet wordt de ADAR-sequentie (oranje) afgelezen dat weer zorgt enz. Inmiddels is de ADAR-sequentie in het circuit-RNA vervangen door de veel kleinere MCP-ADAR-sequentie (afb: Wyssinstituut)
Darth Vader is een figuur uit de Star Wars-films. Onderzoekers van het Wyssinstituut in de VS moeten daar bij de naamgeving van hun methode (DART VADAR) ongetwijfeld aan gedacht hebben. Met die RNA-techniek zouden RNA-therapieën gerichter hun werk kunnen doen zonder over de schreef te gaan. De techniek zorgt er voor dat het aan cellen toegevoegde materiaal pas ‘in actie’ komt in afhankelijkheid van de omstandigheden in de cellen.
De onderzoekers veranderden een natuurlijk enzym en gebruikten ‘sensoren’ die reageren op de aanwezigheid van een bepaalde RNA-verbinding in de cel om de translatie (omzetting in eiwitten) van de genetische lading die aan de cel is toegevoegd in gang te zetten. Met DART VADAR zou de reikwijdte van RNA-technieken en van RNA-therapieën kunnen worden uitgebreid denken de onderzoekers.
RNA-vaccins zijn natuurlijk sedert de coronapandemie gemeengoed geworden, maar voor veel ziektes heb je daar niet veel aan want die vaccins zijn gericht op ziekteverwekkers die zich door het hele lichaam kunnen verspreiden. Bij de bestrijding van kankers of erfelijke ziektes gaat het vaak om bepaalde organen of celtypen.
Er zijn methoden ontwikkeld om met behulp van RNA’s therapieën te ontwikkelen die alleen ‘in actie’ komen als er bepaalde stoffen in de cel aanwezig zijn. Die schijnen zelfs ‘slimme’ therapieën genoemd te worden.
Het probleem is dat je dan elke keer iets nieuws moet verzinnen, een nieuwe ‘sensor’ maken, om dat kunstje voor elkaar te krijgen. De onderzoekers stellen nu een methode ontwikkeld te hebben die het makkelijk maakt om die ‘slimme’ therapieën te ontwikkelen.
De onderzoekers rond Jim Collins noemden hun systeem dus DART VADAR, kort voor Detection en Amplification of RNA Triggers via ADAR (ontdekking en versterking van RNA-triggers via ADAR, waarbij dat laatste weer slaat op adenosinedeaminases (enzymen) die werken op RNA). Ze gebruikten daarbij een enzym dat werkt op RNA (adenosinedeaminase; as).
Daardoor zijn ze in staat om er voor te zorgen dat een afgeleverd genmateriaal (eigenlijk een gensequentie gecodeerd in RNA) pas uitgelezen in het ribosoom wordt als er een bepaalde stof of omstandigheid is gedetecteerd, afhankelijk van ziekte of celtype. In dit verband spreekt Collins over ‘circuits’. “Met deze RNA-circuits kun je behandelingen programmeerbaar maken afhankelijk van celtype en toestand van de cel, terwijl je de onbedoelde effecten minimaliseert”, zegt hij.
“Ons idee was de elementen van een responsieve RNA-sensoren zoals meting en activering te ontkoppelen. Dan wordt het makkelijker om dergelijke circuits voor nieuwe doeleinden te ontwikkelen. Idealiter wilden we het genmateriaal (de RNA-sequentie) kunnen veranderen zonder elke keer de sensor te hoeven veranderen”, zegt medeonderzoeker Raphaël Gayet.
ADAR
ADARs zijn enzymen die het nucleotide adenosine bij dubbelstrengige RNA-moleculen verandert in inosine. Die omzetting maakt de dubbele RNA-streng instabiel. Dat zou een normale reactie zijn van cellen op een virusaanval.
De onderzoekers dachten dat ze die natuurlijke reactie zouden kunnen gebruiken om een RNA-sensor te maken. Dus ontwierpen ze een enkelstrengig RNA met verschillende ‘modules’: een celspecifieke sequentie, een stopcodonsequentie (UAG) in het midden en een sequentie die codeert voor een fluorescerend eiwit.
De stopcodon zorgt ervoor dat geen van de eiwitsequenties erna uitgelezen worden. Zo’n nucleotidevolgorde is voor het ribosoom waar de eiwitten worden aangemaakt het sein om de productie te staken. Als dat enkelstrenging RNA-molecuul zich nu bindt aan een complementair RNA dan krijg je dus een dubbelstrengig molecuul.
Nu wordt het even doorbijten. Dit stukje gaat over al of niet complementaire nucleotiden, de bouwstenen van DNA en RNA. De onderzoekers maakten hun circuit (sensor-RNA) zo dat de A (=adenosine) van de stopcodon niet ‘past’ op de C (cytosine) van het doel-RNA (dat zou eigenlijk een U=uracil moeten zijn).
Daardoor kan die A-nucleotide gevonden worden door de ADAR en omgezet worden in een I (=inosine). Daarmee is UAG niet langer een stopcodon en kan het RNA verder worden afgelezen in het ribosoom. Het fluorescerende eiwit wordt dan ook aangemaakt en de cel is zichtbaar actief. Die toevoeging is onnut voor klinische behandeling, maar zou vervangen kunnen worden door een sequentie van een aan te maken (helend) eiwit.
Voorlopig moest er nog wel wat aan dat systeem gesleuteld krijgen om het klinisch praktisch te maken. Zo was de ADAR-sequentie veel te groot zijn om die met kreupel gemaakte virussen af te kunnen leveren in cellen. Dus sleutelden de onderzoekers wat aan het enzym, waardoor ze een eiwit(sequentie) kregen dat (die) veel kleiner was: MCP-ADAR. Ze veranderden ook de ADAR-sequentie in hun RNA-sensor zodat die codeerde voor MCP-ADAR.
Als nu de RNA-sensor werd geactiveerd door een natuurlijke ADAR in de cel dan kon het geproduceerde MCP-ADAR zich binden aan andere sensoren om ervoor te zorgen dat er nog meer MCP-ADAR wordt aangemaakt (enz, de versterking in de naam).
P53
Om de nieuwe versie uit te proberen keken ze of ze ermee een puntmutatie (een enkele veranderde nucleotide in een gensequentie) konden vinden in de p53-tumoronderdrukker-gen voor mensen. Die proef slaagde.
Vervolgens keken ze of ze met hun systeem ook een onderscheid konden maken tussen de diverse ontwikkelingsstadia van gezonde cellen. Ook die proef slaagde. Nu afwachten of en wat die techniek kan betekenen voor het bestrijden van ziektes.
Bron: Science Daily
