De structuur van RNA-polymerase II (afb: wikiMedia Commons)
Als we op school geweest zijn dan weten we dat ons leven is vastgelegd in een 2 m lang molecuul: DNA. De instructiedelen van DNA (=genen) worden afgelezen en overgebracht op RNA dat op zijn beurt weer wordt afgelezen om eiwitten te vormen, de werkpaarden in wat leven heet. Een belangrijke rol in die dans speelt het enzym RNA-polymerase II (Pol II)
Het is bekend dat Pol II zich langs het gen moet voortbewegen in perfecte harmonie met andere biologische processen. Afwijkingen in de beweging van dit enzym zijn in verband gebracht met kanker en veroudering, maar technische obstakels verhinderden het om precies te bepalen hoe deze belangrijke moleculaire machine zich door het DNA beweegt en wat de pauzes en versnellingen ervan regelt. Een nieuwe studie zou veel van de kennishiaten invullen, aldus de betrokken onderzoekers.
Ze ontwikkelden een een-molecuulplatform om individuele transcriptiecomplexen van zoogdieren aan het werk te bekijken. Het resultaat is een beeld van hoe deze moleculaire machine versnelt, pauzeert en schakelt terwijl hij genetische informatie overschrijft.
“Wat echt opvallend is, is hoe deze machine bijna functioneert als een nauwkeurig afgesteld apparaat”, zegt Shixin Liu van de Rockefelleruniversiteit. “Het heeft het equivalent van verschillende versnellingen of snelheidsmodi, elk aangestuurd door de binding met verschillende regulerende eiwitten. We hebben voor het eerst ontdekt hoe elke versnelling wordt aangestuurd.”
“We zien eindelijk waar Pol II zich bevindt, zowel in tijd als ruimte, tijdens het proces”, zegt medeonderzoeker en inspirator Joel Cohen. “Ons platform stelde ons in staat om goed te beoordelen wanneer deze machine schakelt en hoe snel hij gaat.”
Pol II, al meer dan 50 jaar geleden voor het eerst ontdekt door Robert Roeder van Rockefeller, beweegt zich stap voor stap langs het DNA-molecuul en bouwt een bijpassende RNA-streng op die uiteindelijk eiwitten zal opleveren. Pol II reist echter niet met een constante snelheid door het DNA, vooral niet bij hogere organismen zoals de mens.
Na de initiatie vertraagt het en pauzeert het vaak rond het begin van een gen, voordat regulerende eiwitten zoals P-TEFb en PAF1C het in een snelle transcriptiemodus brengen. Naarmate het einde van een gen nadert, vertraagt het enzym weer om het netjes af te ronden.
Dit tempo is cruciaal. Gaat het te snel of te langzaam dan kunnen de RNA-moleculen niet goed worden verwerkt of gecoördineerd met andere vitale celprocessen. Fouten in de snelheidsregeling van Pol II zijn in verband gebracht met veroudering en een groot aantal ziekten, waaronder kanker.
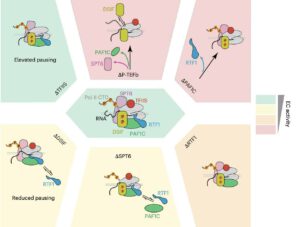
Het aflezen van DNA en ‘overschrijven’ op RNA gaat gepaard met tempowisselingen ‘aangezwengeld’ door verschillende eiwitten (afb: Shixin Liu et al./Nature Structural & Molecular Biology)
Liu: “Veel mensen zijn zich niet bewust van deze verbanden. Sterker nog, mij wordt vaak gevraagd: zolang het RNA kan maken en we weten hoe het dat doet, maakt het ons dan echt uit hoe snel de machine is, of dat die soms pauzeert? Dat is belangrijk juist omdat we weten dat de kinetiek van transcriptie wezenlijk is voor een juiste genexpressie en ook verband houdt met verschillende ziekten.”
Beperkingen
Technische beperkingen verklaren grotendeels waarom eerdere studies moeite hadden om ondubbelzinnig te verhelderen hoe Pol II zijn beweging regelt. Technieken die gemiddelden over verschillende moleculen maten, vertroebelden de bijdragen van individuele eiwitten, terwijl studies met afzonderlijke moleculen in eenvoudiger organismen zoals gist de complexe regelmechanismen van zoogdiercellen niet volledig weerspiegelden.
Liu, deskundig in methoden met afzonderlijke moleculen, realiseerde zich dat hij deze barrières alleen kon overwinnen door een transcriptiesysteem van zoogdieren in vitro na te bouwen, stukje bij beetje uit gezuiverde eiwitten en dit te combineren met geavanceerde beeldvormingstechnieken en algoritmen.
Het idee kreeg vaart door een toevallige ontmoeting met Cohen. “We zaten samen en Joel vroeg me of ik aan iets interessants werkte”, herinnert Liu zich. “Ik heb hem dit project voorgesteld. We zijn er samen naar gaan kijken en sindsdien werken we samen. Ik ben zo blij dat we die dag aan dezelfde tafel zaten.”
Cohen: “Ons werk is een samenwerking tussen verschillende disciplines, onze twee labs en ook over landsgrenzen heen. Als we willen dat de wetenschap floreert, moeten we grensoverschrijdende samenwerking blijven stimuleren.”
Een ‘versnellingsbak’
Samen ontwikkelden de onderzoekers een platform dat biochemie, beeldvorming van afzonderlijke moleculen en berekeningen combineert om Pol II in groot detail aan het werk te zien. Door de transcriptiemachinerie stukje bij beetje te reconstrueren uit gezuiverde zoogdiereiwitten en de beweging ervan direct te volgen – en door een computationeel raamwerk en structurele modellering te gebruiken om precies te bepalen wanneer het enzym van versnelling verandert – toonden de onderzoekers aan dat verschillende belangrijke regulerende eiwitten de beweging van Pol II bepalen.
Zo is P-TEFb, een soort hoofdschakelaar, fosforyleert zowel Pol II als een eiwitcomplex, DSIF, om de volledige activiteit ervan te ontsluiten. DSIF bleek ingewikkelder dan verwacht. Afhankelijk van de toestand kon het Pol II versnellen of of afremmen.
Uiteindelijk bleek het eiwit PAF1C de belangrijkste versneller van Pol II te zijn, die de transcriptie in gang zette zodra het DNA eraan was gebonden. Het SPT6-eiwit speelde de rol van stabilisator en zorgde ervoor dat PAF1C stevig vastzat, zodat de machine soepel kon blijven draaien.
Zodra PAF1C op zijn plaats zat, zorgde dat eiwit ervoor dat nog een andere factor, RTF1, zich kon binden. RTF1 zorgde voor een extra impuls voor de afleessnelheid en schakelde Pol II in een hogere versnelling, een stap waarvoor PAF1C absoluut nodig was, maar DSIF niet. Dit suggereerde een functioneel verband tussen PAF1C, RTF1 en DSIF in zoogdiercellen dat nog niet in gist is waargenomen, wat op de evolutionaire verfijning van het systeem wijst.
“Dit is de eerste keer dat we Pol II bij zoogdieren direct met een fysiologische snelheid hebben kunnen zien bewegen”, zegt zegt Yukun Wang. Omdat we de verschillende verlengingsfactoren hebben gemarkeerd, konden we ook hun bindingskinetiek meten tijdens de feitelijke transcriptie. Soms is zien begrijpen. De kracht van directe verbeelding is dat je het molecuul echt kunt zien – je kunt kijken en leren.”
Verstoringen
De bevindingen werpen ook licht op hoe verstoringen in deze sturing kunnen bijdragen aan kanker en veroudering door nieuwe inzichten te bieden in factoren zoals P-TEFb. Dat eiwit wordt beschouwd als een veelbelovend doelwit voor medicijnen tegen leukemie en vaste tumoren.
Liu: “P-TEFb is notoir moeilijk te remmen zonder toxische bijwerkingen. Ons werk kan aanknopingspunten bieden voor het ontwerpen van specifiekere therapieën. Tegelijkertijd zou inzicht in hoe Pol II door elk van de stadia in gezonde cellen wordt gereguleerd ons een nog breder begrip moeten geven van wat er mis kan gaan bij ziekte.”
Het grootste effect van de studie is echter mogelijk het platform zelf, denken de onderzoekers. Door te bewijzen dat visualisatie van afzonderlijke moleculen mogelijk is in een volledig gereconstrueerd zoogdiersysteem, hebben ze een middel ontwikkeld dat nu kan worden gebruikt om langlopende vragen in de biologie te beantwoorden. Er wordt al gewerkt aan de verbetering van het platform door nucleosomen toe te voegen, de basiseenheden van DNA-verpakking in eukaryotische chromosomen, om beter te begrijpen hoe Pol II zich door sjablonen beweegt die meer lijken op zijn natuurlijke omgeving.
De mogelijke toepassingen van het rekendeel van het platform kunnen ook verreikend zijn. “Alles wat te maken heeft met navigatie in de ruimte en snelheidsveranderingen zou potentieel gebruik kunnen maken van deze programmauur”, stelt Cohen.
Bron: phys.org
